J’aime bien la biologie, depuis ma tendre enfance. Je pensais même pouvoir un jour pouvoir faire de la bioinformatique mais j’ai vite été lassée des études en fac, donc j’ai dû faire un peu une croix sur ce projet.
Then, I left to Marseille, tout ça …
Mais dernièrement, j’ai eu pas mal de remous professionnels, quitté la dernière entreprise où j’étais en CDI (plutôt rageusement). J’ai donc passé quelques mois à construire des relations avec des clients, privilégiant l’approche freelance, et j’ai également trouvé un projet perso qui puisse m’occuper et approfondir mes connaissances en Symfony. Parce que depuis Spark, j’ai horreur d’être inoccupée. Et puis quand mon chat est mort, il a fallu un échappatoire pour penser à autre chose. Et puis j’avais une revanche perso à relever (n’est-ce pas, Guillaume ?).
J’ai trouvé donc le projet BioPHP (qui est sans URL Fixe, y’a un site ici aussi), cherchant une librairie de bioinformatique en PHP, par curiosité après avoir vu que des librairies du genre existaient en GO. Et … comment dire ? Voir des 2003, dernières mises à jour en 2009 peut-être, des fonctionnalités en PHP4, du HTML, du PHP, des data mélangées dans le code. Aucun design pattern, du code spaghetti, des fonctionnalités qui se retrouvent un peu partout. Ouch. Pourtant les fonctionnalités sont intéressantes et offrent du potentiel, les algorithmes sont hyper poussés. Cette librairie a été développée par d’excellents biologistes qui ne sont pas familiarisés avec le dev. D’ailleurs en poussant la recherche, le projet est critiqué et mal noté.
L’idée de départ des gars était super sympa : intégrer une librairie pour créer des applis de bioinformatique (plutôt orientées génétique) en PHP, comme il en existe en PERL, Go, Python … Mais il est vrai qu’à l’époque, à moins de télécharger bêtement et utiliser les fonctions require, on ne pouvait pas faire grand-chose d’autre. A part peut-être les intégrer dans PEAR ou PECL, chose que je n’ai jamais faite, donc je ne connais pas le process. PHP était vraiment à l’époque le vilain petit canard du monde des langages, et c’est à ce niveau qu’on voit son évolution fulgurante. Car aujourd’hui les outils ont évolué, nous avons les frameworks, Composer, Packager, Github, les tests unitaires et fonctionnels … en un coup de « composer install », on peut intégrer une librairie.
Comment procéder quand on est face à ce genre de challenges ? A vrai dire je n’ai pas vraiment établi de plan d’action dès le départ, peut-être une mauvaise idée, mais je me suis dit que c’était un projet pour lequel j’avais tout mon temps, et le droit à l’erreur. Car ce projet pris sur mon temps perso a été commencé en février dernier. En gros voici comment je suis en train de procéder :
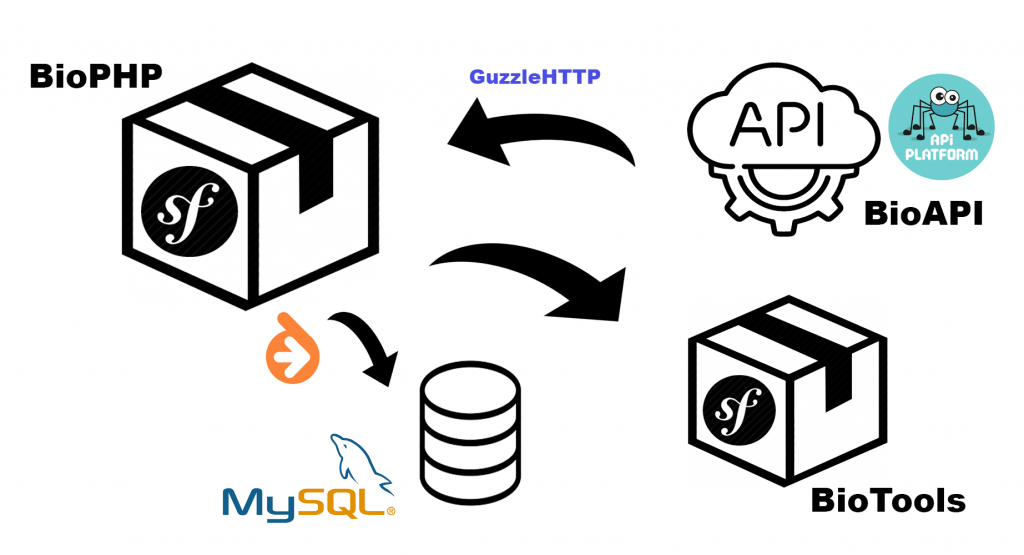
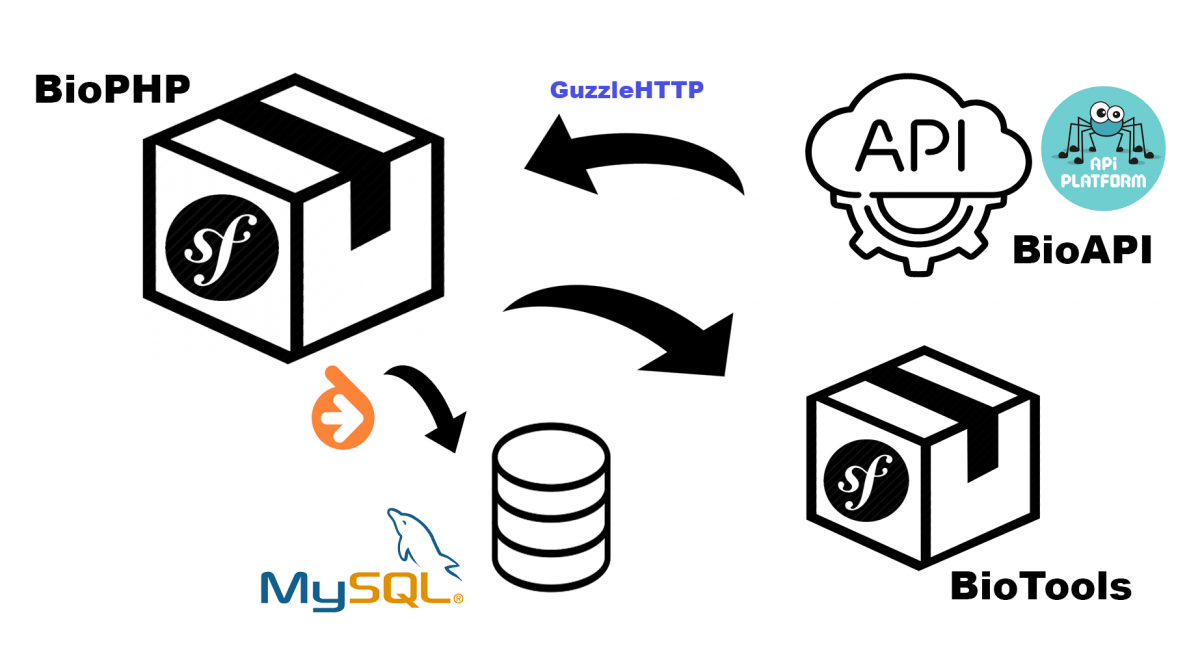
- Installation de Symfony, création de 3 bundles : AppBundle (core de l’appli, MinitoolsBundle (qui intègre des outils qui pouvaient utiliser la librairie), et DatabaseBundle, qui m’a servi de « bundle temporaire » pour créer mes classes entités, raccordées avec Doctrine (parce que franchement, la classe seq.php … non, non, et non).
- Premier coup de machette dans le code. Nettoyage des fonctionnalités, renommage des variables suivant leur type, restructuration des classes originales. Séparation des classes-entités de ce qui sera intégré dans des services.
- Mise de côté des data. D’abord j’avais pensé à les inclure en YAML dans des paramètres, très mauvaise idée. Base de données ? Je voudrais plus la réserver à l’appli, pour moi la BDD ne doit servir qu’au stockage des séquences. Reste une idée sympa, les intégrer dans une API, ce que je voulais construire depuis longtemps pour utiliser API-Platform, un outil performant qui permet de pouvoir créer facilement ses propres API Rest. Le plus : on peut créer une couche interface qui permettra de créer un adapter, et donc de changer d’API, au cas où, tout en implémentant le modèle requis. Même si le CRUD n’est pas à 100% respecté. Les données de l’API ne sont pas destinées à être modifiées, dans l’immédiat.
- Refacto des « Minitools » pour bien m’imprégner de la logique de ces outils, création de l’interface web en TWIG, des services correspondants : je repère les duplicatas, crée de nouveaux services qui pourront être utiles dans le core. Car à la base, ces tools devaient intégrer les fonctionnalités de BioPHP. De mon côté ils me permettent aussi de me replonger dans les fondamentaux de la biologie : qu’est-ce qu’une protéine ? un acide aminé ? un enzyme etc …
- Refacto en test manuel : depuis le temps, les fonctionnalités sont en friche, donc je les fais remarcher, petit à petit, en continuant le labeur de nettoyage et de refacto. Je repère ainsi les fonctionnalités qui seront intégrées dans les « Minitools ». Cette partie sera désolidarisée (renommée en BioTools, pour caler au nommage général) du package à terme, pour des raisons logiques : le multi-repo c’est le bien, je vous dis.
- Tests unitaires du core : maintenant que je sais que ça marche, je repasse en mode « semi-TDD » et intègre les tests unitaires. Car un gros travail de refacto m’attend : la définition des différents design patterns de AppBundle. -> je suis ici \o/ .
- Découpage du projet pour en créer un projet Packagist. Et certainement montée en version de Symfony, redéfinition des dépendances d’injection etc …
- Création des tests unitaires et fonctionnels (avec Behat) de BioTools, refacto et envoi à Packagist.
Au prochain épisode, je rentrerai plus en détails sur la refacto d’AppBundle et des design patterns choisis.
Ha oui, et sinon, toutes les infos du projet sont là … Amelaye’s BioPHP.