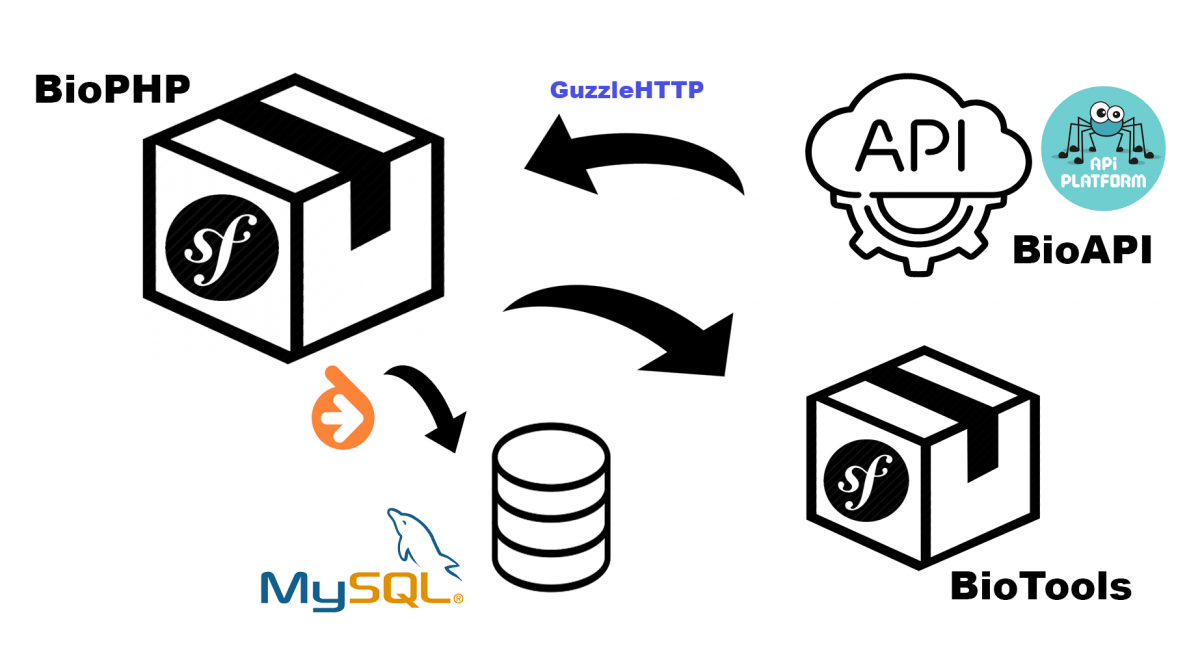
Le souci de la librairie d’origine de BioPHP, c’est son absence de design pattern. Le code d’origine prévoyait d’inclure deux systèmes de bases de données (Genbank et Swissprot en l’occurence) voire d’autres, car j’ai vu une dizaine d’autres formats.
Or le but du jeu c’est d’avoir une structure assez carrée pour pouvoir ensuite manipuler les données.
Tout se joue dans le fichier d’origine seqdb.php, je vous laisse regarder la tambouille. Dans ce cas-là, il faut surtout comprendre la logique, la garder et tout casser en même temps. A coups de massue.
La première des choses que j’ai faite, c’est compartimenter une classe par fichier, et un fichier par classe : donc on a trois fichiers, SeqDB, et des fonctions parse_swissprot et parse_id que je ramène dans une classe chacune, et que je renomme au passage car sinon on n’y comprend pas grand-chose. Je passe l’étape de « nettoyage », remplacement des fonctionnalités obsolètes, découpage du code en méthodes privées, simple, basique.
Deuxièmement, c’est d’utiliser un Iterator pour le flux des fichiers car le script réinvente la roue avec les next() et les prev().
Ensuite j’ai refactorisé en gardant l’ancienne structure de seq.php, pour arriver à comprendre petit à petit la logique, à quoi correspond chaque champ du fichier qui est envoyé. Avant le grand coup d’envoi, puisque les structures qui correspondent aux séquences vont être factorisées à leur tour, pour cela je me suis inspirée du modèle de données qui avait été proposé par l’équipe, en y mettant deux trois ajustements :
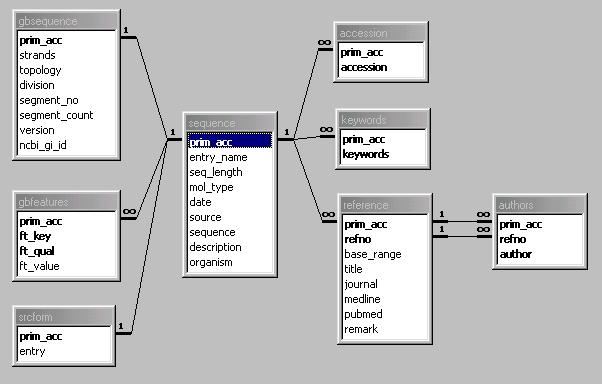
Une fois que c’est établi, que ça marche et parse, il faut intégrer ce système au parsing des fichiers tout en permettant à l’utilisateur de lui simplifier un maximum le travail.
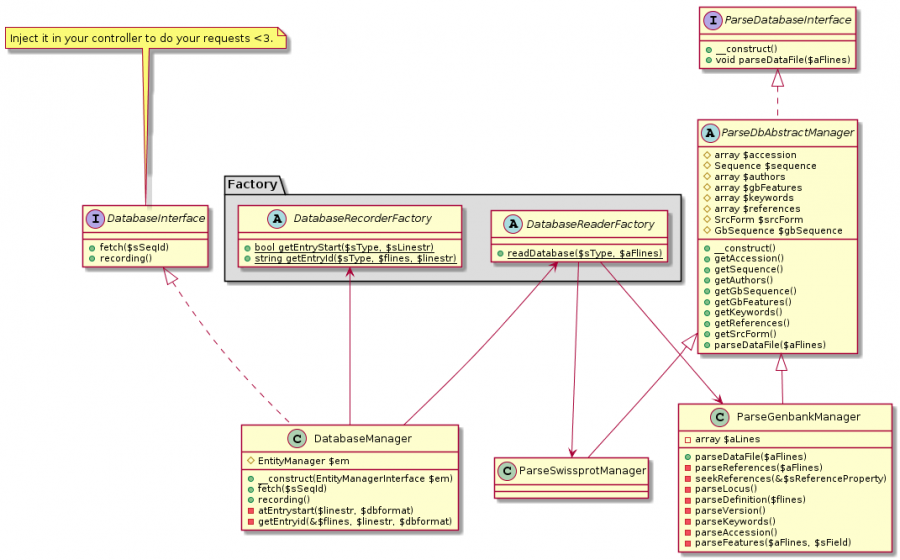
Il fera appel à une interface. Une seule. Et la librairie se chargera du reste. Voici comment (le travail n’est pas terminé ce qui explique pourquoi ParseSwissProt est vide mais c’est en cours) :
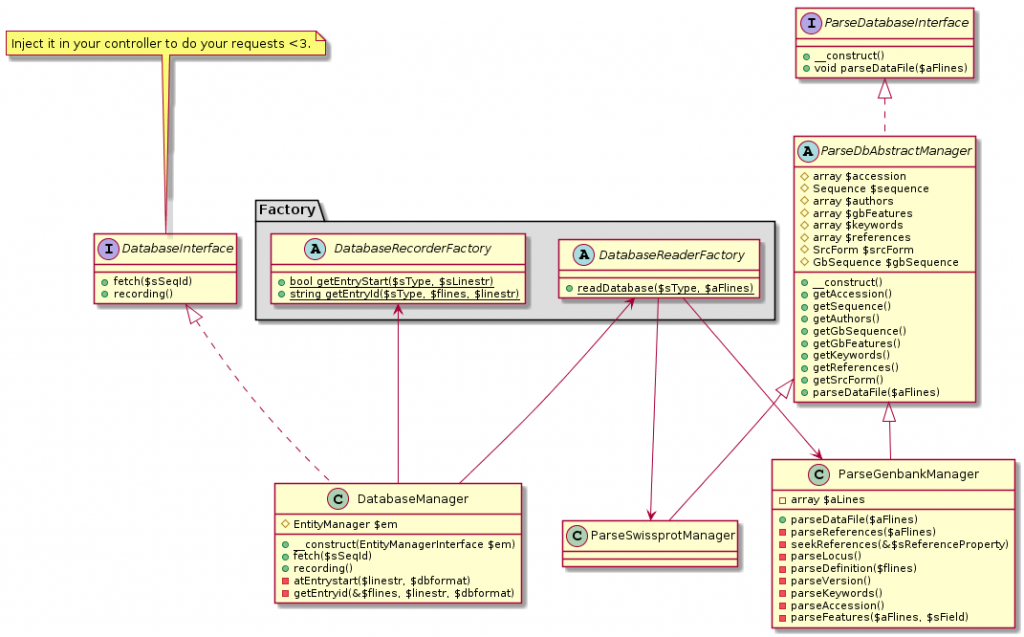
Utiliser l’interface DatabaseInterface fera appel via l’injection de dépendances à DatabaseManager qui se charge de vérifier si le fichier existe en base, ou d’aller chercher de la donnée. Suivant le cas de figure, ce sont des factory qui seront appelées et qui joueront les aiguilleurs en appelant les services correspondants. Ces services « final class » ont une notion d’héritage sur un autre service, abstrait, ParseDbAbstractManager, qui contient l’architecture de la donnée qui sera renvoyée dans le controller de l’utilisateur.
Ce qui permet dans tous les cas de renvoyer une donnée, qui, quel que soit le schéma de départ, sera uniforme. Et ce grâce à deux design pattern : Factory et Abstract.
Du coup, si on veut ajouter une autre structure de fichier, il suffira de modifier les factory et d’ajouter un service qui sera chargé uniquement de parser la donnée. Ceci dit, je pense qu’il est même possible de passer outre la modification des factory, je dois réfléchir sur ce point.
Je crois que cette partie est la plus complexe de mon projet, car je ne suis pas familiarisée avec les bases de données textuelles propres à la bioinformatique, et le script de départ contenait pas mal de bugs. Du coup si des biologistes chevronnés constatent des bugs à l’utilisation, ils peuvent me contacter, ce sera volontiers que je procèderai aux ajustements.